PLM重大突破!上海交大与上海AI Lab最新成果入选NeurIPS 24,ProSST有效整合蛋白质结构信息
AIGC动态欢迎阅读
原标题:PLM重大突破!上海交大与上海AI Lab最新成果入选NeurIPS 24,ProSST有效整合蛋白质结构信息
关键字:结构,蛋白质,模型,序列,解读
文章来源:HyperAI超神经
内容字数:0字
内容摘要:
作者:田小幺
编辑:十九,李宝珠
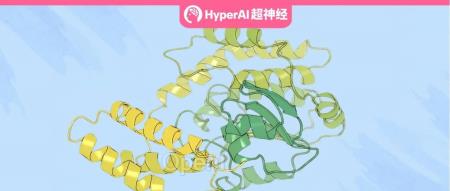
上海交通大学自然科学研究院/物理天文学院/张江高研院/药学院洪亮教授课题组,上海交大助理研究员周冰心,联合上海人工智能实验室青年研究员谈攀,成功开发出具有结构感知能力的预训练蛋白质语言模型 ProSST,能够将蛋白质结构和氨基酸序列信息有效融合,从而在监督学习任务中显著超越现有模型。蛋白质作为生命体的关键分子,其序列决定结构,结构决定功能,蛋白质的功能与其三维结构紧密相关。数十年来,科学家们利用 X 射线晶体学、核磁共振等技术,解析了数千种蛋白质结构,为人们理解蛋白质功能提供了重要线索。然而,面对数百万级别的蛋白质数量,解析所有蛋白质结构的任务显得异常艰巨。
受自然语言处理领域中预训练语言模型的启发,预训练的蛋白质语言模型 (PLMs) 应运而生。通过在海量未标记的蛋白质序列数据上进行学习,PLM 能够捕捉到蛋白质序列的复杂模式和相互作用,这为蛋白质功能的预测、结构分析以及蛋白质-蛋白质相互作用的识别带来了性的进步。
然而,大多数 PLM 主要关注蛋白质序列建模,忽略了结构信息的重要性,这主要是因为结构数据的缺乏。随着 AlphaFold 和 Ro
原文链接:PLM重大突破!上海交大与上海AI Lab最新成果入选NeurIPS 24,ProSST有效整合蛋白质结构信息
联系作者
文章来源:HyperAI超神经
作者微信:
作者简介:
© 版权声明
文章版权归作者所有,未经允许请勿转载。
相关文章
暂无评论...


 粤公网安备 44011502001135号
粤公网安备 44011502001135号